10.3 安装包
R出厂时被设置为访问CRAN的包库(提示选最近的镜像),如运行Windows则会访问CRANextra。CRANextra包含一些在Windows下构建时需特别注意的包,它们不能在通常的CRAN服务器上托管。要访问其他存储库,输入setRepositories (),选择您想要的库。图10-2显示了它的可选项。
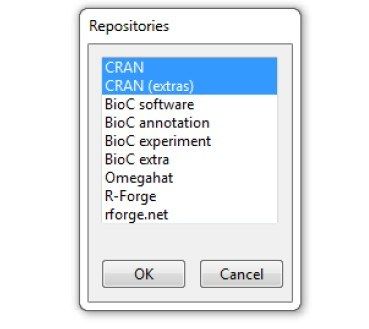
图10-2:可用的包库列表
Bioconductor包含了与基因组学和分子生物学的包,而R-Forge和RForge.net大多包含了开发中的版本,它们最终出现会在CRAN。使用available.packages可以查看在库中的包的所有信息(请注意,因为那里的包有成千上万个,所以这需要好几秒钟运行):
View(available.packages())
除了这些库,还能从网上很多其他的储存库中找到R的包文件,如GitHub、bitbucket和Google Code。正如下文所讨论的那样,从GitHub上取包特别容易。
许多IDE也提供了点选式的方法来安装包。 在R的GUI中,在Package菜单中提供了“Install Package(s) …”的选项从资源库中安装包,以及使用“Install package(s) from local zip files…”来从你已下载的包中安装。图10-3显示R的GUI菜单。
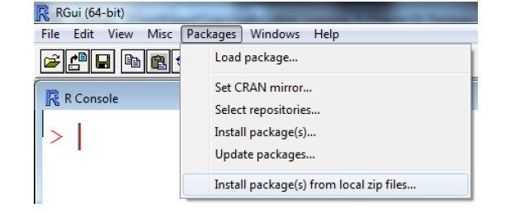
图10-3: 使用R的GUI来安装包
你也可以使用install.packages函数来安装包。不使用任何参数将弹出同样的GUI界面,就像你点击了“Install package(s) …”菜单选项一样。通常你会想指定要安装的包的名字以及要访问的存储库的URL地址。在CRAN主站http://cran.r-project.org/mirrors.html上可以查到所有CRAN镜像的RUL列表。
此命令将(尝试)下载时间序列分析软件包xts、zoo和它们所有依赖的包,然后把它们安装到默认的库位置(.libPaths返回的第一个值):
install.packages(c("xts", "zoo"),repos = "http://www.stats.bris.ac.uk/R/")
如果想安装到不同的位置,把参数lib传递给install.packages:
install.packages(c("xts", "zoo"),lib = "some/other/folder/to/install/to",repos = "http://www.stats.bris.ac.uk/R/")
很明显,你需要连上互联网才能使R下载包,而且你需要有把文件写入库目录的权限。在企业的内部网中,R的互联网访问可能会受限。在Windows下,你可以让R使用internet2.dll来访问因特网,就像使用IE浏览器一样来绕过访问限制。要做到这一点,请输入:
setInternet2()
如果以上的方法都不可行,访问http:// web/packages/available_packages_by_name.html并手动下载你所需的包(以及所有依赖包),然后安装这些tar.gz/tgz/zip文件:
install.packages("path/to/downloaded/file/xts_0.8-8.tar.gz",repos = NULL, # repo为NULL意味着包已经被下载type = "source" # 这意味着现在就构建包)install.packages("path/to/downloaded/file/xts_0.8-8.zip",repos = NULL, # 还需要这个type = "win.binary" # 只是Windows!)
要直接从GitHub上安装包,你首先要安装devtools包:
install.packages("devtools")
install_github函数接受GitHub库的名称,此库将包含你所需要的包(通常与包本身的名称一样),以及维护此库的作者名。例如,要得到开发版本的reporting包knitr,输入:
library(devtools)install_github("knitr", "yihui")
