12.3 读取文本文件
有众多的格式和文本文件标准可用于存储数据。用于存储数据的通用格式为分隔符值(即CSV或制表符分隔文件)、可扩展标记语言(XML)、JavaScript对象表示法(JSON)和YAML(这是YAML Ain't Markup Language的递归表示)。文本数据的其他来源结构会比较松散,例如书本,其中并没有包含任何正式的文本数据(即标准化和可被机器解析的)结构。
将数据存储在文本文件中的主要优点是:它们可被几乎所有的其他数据分析软件或人读取。这使得你的数据能广泛地被重用。
12.3.1 CSV和制表符分隔(Tab-Delimited)文件
矩形(类似电子表格的)数据通常存储在带有分隔符的文件中,特别是逗号分隔值(CSV)和制表符分隔值文件。read.table函数将读取这些分隔符文件,并将结果存储在一个数据框中。它就是这么简单,只需输入文本文件的路径,即可导入其内容。
RedDeerEndocranialVolume.dlm是一个以空格符分隔的文件,它包含了一些使用不同技术测量得到的马鹿的颅容积数据。(对于那些对鹿头骨感兴趣的人来说,该方法就是计算机X射线断层术,它会用到Finarelli方程。它用玻璃珠填充满头骨,然后使用卡钳测量其长度、宽度和高度。在某些情况下,作第二次测量能够知道所采用技术的准确性。我已确认:鹿死很久之后才会将它们的头骨填满珠子!)数据文件可以在learningr包的extdata文件夹中找到。前几行数据请参见表12-1 。
表12-1: RedDeerEndocranialVolume.dlm样本中的数据
| SkullID | VolCT | VolBead | VolLWH | VolFinarelli | VolCT2 | VolBead2 | VolLWH2 |
|---|---|---|---|---|---|---|---|
| DIC44 | 389 | 375 | 1484 | 337 | |||
| B11 | 389 | 370 | 1722 | 377 | |||
| DIC90 | 352 | 345 | 1495 | 328 | |||
| DIC83 | 388 | 370 | 1683 | 377 | |||
| DIC787 | 375 | 355 | 1458 | 328 | |||
| DIC1573 | 325 | 320 | 1363 | 291 | |||
| C120 | 346 | 335 | 1250 | 289 | 346 | 330 | 1264 |
| C25 | 302 | 295 | 1011 | 250 | 303 | 295 | 1009 |
| F7 | 379 | 360 | 1621 | 347 | 375 | 365 | 1647 |
该数据有标题行,所以我们需要给read.table传递参数header = TRUE。因为并不是每次都会进行第二次测量,所以不是所有行都是完整的。给read.table传递参数fill = TRUE会使用NA值来代替那些缺失的域。下例中的system.file函数用于定位包中的文件(在下例中,RedDeerEndocranialVolume.dlm文件在learningr包中的extdata文件夹中)。
library(learningr)deer_file <- system.file("extdata","RedDeerEndocranialVolume.dlm",package = "learningr")deer_data <- read.table(deer_file, header = TRUE, fill = TRUE)str(deer_data, vec.len = 1) #vec.len改变了输出的数量## 'data.frame': 33 obs. of 8 variables:## $ SkullID : Factor w/ 33 levels "A4","B11","B12",..: 14 ...## $ VolCT : int 389 389 ...## $ VolBead : int 375 370 ...## $ VolLWH : int 1484 1722 ...## $ VolFinarelli: int 337 377 ...## $ VolCT2 : int NA NA ...## $ VolBead2 : int NA NA ...## $ VolLWH2 : int NA NA ...head(deer_data)## SkullID VolCT VolBead VolLWH VolFinarelli VolCT2 VolBead2 VolLWH2## 1 DIC44 389 375 1484 337 NA NA NA## 2 B11 389 370 1722 377 NA NA NA## 3 DIC90 352 345 1495 328 NA NA NA## 4 DIC83 388 370 1683 377 NA NA NA## 5 DIC787 375 355 1458 328 NA NA NA## 6 DIC1573 325 320 1363 291 NA NA NA
注意,每个列的类已自动确定,行和列的名字也已自动分配。列名(默认情况下)必须是有效的变量名(通过使用make.names),如果不提供行名那么行将就会按1、2、3编号,以此类推。
有很多参数可以用来指定如何读取该文件,其中最重要的可能是sep,它决定了使用哪个字符作为字段之间的分隔符。nrow可以指定读取数据的行数,而skip决定跳过文件开始的多少行。更多高级选项包括:覆盖默认的行名、列名和类,指定输入文件的字符编码,以及输入的字符串格式的列如何声明。
有几个read.table的包装函数使用起来比较方便。read.csv分隔符默认设置为逗号,并假设数据有标题行。read.csv2是它的欧洲表亲,它使用逗号作为小数位,并用分号作为分隔符。同样地,read.delim和read.delim2将分别使用句号或逗号作为小数位来导入制表符分隔的文件。
早在2008年8月,在英国的洛斯托夫特,环境、渔业和水产养殖科学中心(CEFAS)的科学家把贴上标签的压力和温度传感器放在一种棕色螃蟹身上,并把它们投入北海。螃蟹自在地游荡了一年之后1,渔民抓住了它,并把标签送回了CEFAS。
1具体就是从北海东部德国附近迁移到北海西部英国附近。
从该标签中获取的数据以及一些元数据都存储在一个CSV文件中。文件的前几行是这样的:
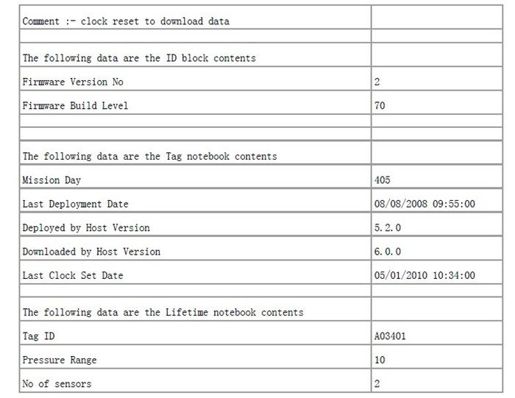
在这种情况下,我们不能仅调用read.csv就把所有东西都读取出来,因为不同的数据块中所含的字段数量不同,而且每个字段也确实不同。我们需要使用read.csv中的skip和nrow参数指定要读取文件中哪些位置:
crab_file <- system.file("extdata","crabtag.csv",package = "learningr")(crab_id_block <- read.csv(crab_file,header = FALSE,skip = 3,nrow = 2))## V1 V2## 1 Firmware Version No 2## 2 Firmware Build Level 70(crab_tag_notebook <- read.csv(crab_file,header = FALSE,skip = 8,nrow = 5))## V1 V2## 1 Mission Day 405## 2 Last Deployment Date 08/08/2008 09:55:00## 3 Deployed by Host Version 5.2.0## 4 Downloaded by Host Version 6.0.0## 5 Last Clock Set Date 05/01/2010 10:34:00(crab_lifetime_notebook <- read.csv(crab_file,header = FALSE,skip = 15,nrow = 3))## V1 V2## 1 Tag ID A03401## 2 Pressure Range 10## 3 No of sensors 2
提示
colbycol和sqldf包中的函数可用于把CSV文件的部分数据读取到R中。如果你并不需要所有列或所有行,这能加快速度。
在导入此类文件时,你可以使用scan函数来真正地从底层进行控制,read.table也是基于此函数实现的。通常情况下,scan应该被避免使用,但它对于处理格式奇怪的和非标准的文件非常有用。
提示
如果你的数据是从另一种语言中导入的,那么可能需要把
na.strings参数传递给read.table。对于从SQL导出的数据,则使用na.strings = "NULL"。对于从SAS或Stata导出的数据,使用na.strings = "."。从Excel中导出的数据,使用na.strings = c("", "#N/A", "#DIV/0!", "#NUM!")。
与此相反的任务是写入文件,这通常比读取文件要更简单,因为你无需担心读取文件时出现的各种怪事——通常人们想创造一些标准的东西。很明显,write.table和write.csv分别对应着read.table和read.csv的读操作。
这两个函数都需要一个数据框和写入文件的路径作为参数。它们还提供了几个选项以自定义输出(例如:是否包含列名,应使用什么样的输出文件字符编码) :
write.csv(crab_lifetime_notebook,"Data/Cleaned/crab lifetime data.csv",row.names = FALSE,fileEncoding = "utf8")
12.3.2 非结构化文本文件
不是所有的文本文件都像定界符文件那样有一个定义良好的结构。如果文件的结构松散,更简单的做法是:先读入文件中的所有文本行,再对其内容进行分析或操作。readLines(注意大写字母L)就提供了这种方法。它接受一个文件路径(或文件连接)和一个可选的最大行数作为参数来读取文件。这里将导入莎士比亚的《暴风雨》的古登堡计划版本:
text_file <- system.file("extdata","Shakespeare's The Tempest, from Project Gutenberg pg2235.txt",package = "learningr")the_tempest <- readLines(text_file)the_tempest[1926:1927]## [1] " Ste. Foure legges and two voyces; a most delicate"## [2] "Monster: his forward voyce now is to speake well of"
writeLines用于执行与readLines相反的操作。它写入文件时需要一个字符向量和文件作为输入参数:
writeLines(rev(text_file), #rev执行向量的相反操作"Shakespeare's The Tempest, backwards.txt")
12.3.3 XML和HTML文件
XML文件被广泛地用于存储嵌套数据。许多标准的文件类型和协议都基于它,例如:用于提供新闻资料的RSS(Really Simple Syndication),用于跨计算机网络传递结构化数据的SOAP(Simple Object Access Protocol),以及网页中常用的XHTML。
R的基本包没有读取XML文件的能力,不过XML包已被某R核心成员开发出来了。现在就安装它吧!
install.packages("XML")
当你导入一个XML文件时,该XML包将提供两种选择以存储结果:一是利用内部节点(即使用C代码来存储对象,这是默认值),或使用R节点。通常,你应使用内部节点来存储,因为这样就能用XPath(马上就会谈到它)来查询节点树。
有几个函数可用于导入XML数据,如xmlParse和其他一些使用稍不同的默认值的包装函数:
library(XML)xml_file <- system.file("extdata", "options.xml", package = "learningr")r_options <- xmlParse(xml_file)
使用内部节点的一个问题是,str和head等汇总函数不能和它们一起使用。要使用R-级的节点,请设置useInternalNodes = FALSE(或使用xmlTreeParse,它会默认设置此项属性):
xmlParse(xml_file, useInternalNodes = FALSE)xmlTreeParse(xml_file) # 作用相同
XPath是一种用于查询XML文档的语言,它能基于某些过滤规则寻找到相应的节点。在下例中,我们将在文档//中到处寻找命名为variable的结点,此结点[]的name属性@包含contains了warn字符串。
xpathSApply(r_options, "//variable[contains(@name, 'warn')]")## [[1]]## <variable name="nwarnings" type="numeric">## <value>50</value>## </variable>#### [[2]]## <variable name="warn" type="numeric">## <value>0</value>## </variable>#### [[3]]## <variable name="warning_length" type="numeric">## <value>1000</value>## </variable>
这种查询在提取网页数据中非常有用。正如你所料,htmlParse和htmlTreeParse是用于HTML页面导入的函数,它们的行为方式基本一样。 XML格式在序列化(也即存储)对象时非常有用,这种格式可被大多数其他软件读取。XML包没有提供序列化的功能,但你可以使用Runiversal包中的makexml函数来完成它。Options.xml文件由以下代码创建:
library(Runiversal)ops <- as.list(options())cat(makexml(ops), file = "options.xml")
12.3.4 JSON和YAML文件
XML的主要问题是它太冗长了,且你需要显式地指定数据的类型(它在默认情况下不能区分字符串和数字),这就使得它更冗长了。如果文件大小很重要(例如,当你要在网络上传输大量数据集时),信息过于冗余就成了问题。
于是,有人发明了YAML和它的子集JSON来解决这些问题。它们特别适合于通过网络传输大量数据集,尤其是数字数据和数组。JSON是Web应用程序彼此之间传递数据的事实标准。
有两个包可用于处理JSON数据:RJSONIO和rjson。在很长一段时间内,rjson都存在性能问题,因此唯一值得推荐的包是RJSONIO。不过,性能的问题现在已经修复,所以它也算是一个备选包。在大多数情况下,你使用哪个包都可以。只有当你遇到格式不正确或非标准的JSON时会看出差别。
在读入不正确的JSON时,RJSONIO一般比rjson更宽容。这是否是好事取决于你所使用的场景。如果你想简单地导入JSON数据,RJSONIO就是最好的选择。如果你想对有问题的JSON数据的保持警觉(或许是你的同事生成的——我肯定,你绝不会生成有问题的JSON),那么rjson就是最好的。
幸好,在这两个包中读取和写入JSON数据的函数名基本相同,所以很容易在它们之间切换。在下例中,双冒号::用于把相同名字的函数从不同的包中分别出来(如果只加载两个包中的一个,就不需要双冒号):
library(RJSONIO)library(rjson)jamaican_city_file <- system.file("extdata","Jamaican Cities.json",package = "learningr")(jamaican_cities_RJSONIO <- RJSONIO::fromJSON(jamaican_city_file))## $Kingston## $Kingston$population## [1] 587798#### $Kingston$coordinates## longitude latitude## 17.98 76.80###### $`Montego Bay`## $`Montego Bay`$population## [1] 96488#### $`Montego Bay`$coordinates## longitude latitude## 18.47 77.92(jamaican_cities_rjson <- rjson::fromJSON(file = jamaican_city_file))## $Kingston## $Kingston$population## [1] 587798#### $Kingston$coordinates## $Kingston$coordinates$longitude## [1] 17.98#### $Kingston$coordinates$latitude## [1] 76.8######## $`Montego Bay`## $`Montego Bay`$population## [1] 96488#### $`Montego Bay`$coordinates## $`Montego Bay`$coordinates$longitude## [1] 18.47#### $`Montego Bay`$coordinates$latitude## [1] 77.92
请注意,RJSONIO把每个城市的坐标简化为一个向量。这种功能可通过simplify = FALSE来关闭,这样产生的对象就和rjson产生的完全一样。
有点讨厌的是,JSON的规范不允许无穷值或NaN值,而且它对缺失数的定义比较模糊。这两个包处理这些值的方式有所不同:RJSONIO把NaN和NA映射为JSON的null,但保留正负无穷;而rjson会把所有这些值都转换为字符串:
special_numbers <- c(NaN, NA, Inf, -Inf)RJSONIO::toJSON(special_numbers)## [1] "[ null, null, Inf, -Inf ]"rjson::toJSON(special_numbers)## [1] "[\"NaN\",\"NA\",\"Inf\",\"-Inf\"]"
因为这两种方法都用于处理备受限制的JSON规范,所以如果你发现需要大量地处理这些特殊数字类型(或想在你的数据对象中加些评论),那么最好还是使用YAML。在yaml包中有两个函数能导入YAML数据:yaml.load接受一个YAML的字符串,并将其转换为一个R对象;yaml.load_file也一样,不过它把输入的字符串作为包含YAML文件的路径处理:
library(yaml)yaml.load_file(jamaican_city_file)## $Kingston## $Kingston$population## [1] 587798#### $Kingston$coordinates## $Kingston$coordinates$longitude## [1] 17.98#### $Kingston$coordinates$latitude## [1] 76.8######## $`Montego Bay`## $`Montego Bay`$population## [1] 96488#### $`Montego Bay`$coordinates## $`Montego Bay`$coordinates$longitude## [1] 18.47#### $`Montego Bay`$coordinates$latitude## [1] 77.92
as.yaml则执行相反的任务,它会把R对象转换为YAML字符串。
