3.2 绘制基因组图谱
除了行人车辆东奔西走的情境以外,我们还发现TSP模型具有某些相当不可思议的应用。其中,TSP在遗传学领域的应用最为引人注目。染色体上的标记是基因组图谱的地标,所以过去十年间,精确定位这些标记一直是研究的热点。
在基因组图谱中,每一条染色体上都按顺序排列了一些标记,相邻两个标记之间的距离也都有估测的数值。这些图谱中的标记实际上是DNA片段,恰好只在整个基因组里出现一次,而且可以通过实验室研究工作准确检测到。研究人员利用了它们的唯一性和可检测性,对各个实验室绘制的物理图谱进行核实、对照和合并。研究的重中之重是要精确了解这些标记在染色体上出现的顺序,TSP正是在这一步大显身手。
可以通过不同方法得到这些标记相对位置的实验室数据,其中有一种非常重要的技术称为辐射杂种细胞作图(radiation hybrid mapping),简称RH作图技术。这种方法分为两步。首先将染色体暴露在高剂量的X射线里,使之受到辐射后断裂成若干片段。然后把这些片段和啮齿目动物的遗传物质组合在一起,形成杂交细胞系,再分析确定标记位置。图3-4是这两步操作的简单示意图。
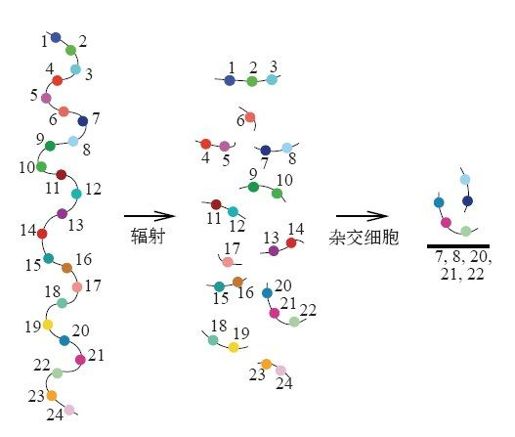
图3-4 辐射杂种细胞作图法原理
RH作图技术的核心思想是,通过分析标记两两之间能否并存于细胞系中,收集总结标记的位置信息。具体说来,如果标记A和标记B在染色体上距离很近,那么辐射就不太可能使染色体在它们之间断开,所以假如在一个细胞系里有标记A,那么很可能也有标记B。反之,如果标记A和标记B在染色体上距离很远,那么就可以预料,A和B将分别出现在不同的细胞系中,两个标记在某个细胞系中并存的可能性极小。研究人员可以巧妙利用这种推理方式,精心得出两个标记间距的实验值。有了两两之间距离的实验值,则测定染色体上标记顺序的问题就转化成为TSP模型。事实上,可以把这一顺序看作周游所有标记的哈密顿通路。这条通路很容易转化为回路,正如我们之前的做法那样,只需要增加一个额外的“虚城市”点即可。
在美国国立卫生研究院(National Institutes of Health),Richa Agarwala和Alejandro Schäffer带领一个小组研究这类基因组问题。针对这些具有实际背景的TSP,他们已经提出解决方法,开发了软件,还考虑到了如何处理错误数据(这属于实验室里的家常便饭)。1他们的软件使用了Concorde代码,保证能找出最优路线。这个软件为许多重要的研究作出了贡献,比如用来构建很多物种的基因组图谱,包括人、恒河猴、马、狗、猫、小鼠、大鼠、奶牛、绵羊和水牛。
1. Agarwala, R., et al. 2000. Genome Research 10, 350–364.
