15.3 探索缺失值模式
在决定如何处理缺失数据前,了解哪些变量有缺失值、数目有多少、是什么组合形式等信息非常有用。本节中,我们将介绍探索缺失值模式的图表及相关方法。最后,如果知道了数据为何缺失,这将为后续深入研究提供许多启示。
15.3.1 列表显示缺失值
你已经学习了一些识别缺失值的基本方法。比如15.2节使用complete.cases()函数列出完整的实例,或者相反,列出含一个或多个缺失值的实例。但随着数据集的增大,该方法就逐渐丧失了吸引力。此时你可以转向其他R函数。
mice包中的md.pattern()函数可生成一个以矩阵或数据框形式展示缺失值模式的表格。将函数应用到sleep数据集,可得到:
> library(mice)> data(sleep, package="VIM")> md.pattern(sleep)BodyWgt BrainWgt Pred Exp Danger Sleep Span Gest Dream NonD42 1 1 1 1 1 1 1 1 1 1 02 1 1 1 1 1 1 0 1 1 1 13 1 1 1 1 1 1 1 0 1 1 19 1 1 1 1 1 1 1 1 0 0 22 1 1 1 1 1 0 1 1 1 0 21 1 1 1 1 1 1 0 0 1 1 22 1 1 1 1 1 0 1 1 0 0 31 1 1 1 1 1 1 0 1 0 0 30 0 0 0 0 4 4 4 12 14 38
表中1和0显示了缺失值模式,0表示变量的列中有缺失值,1则表示没有缺失值。第一行表述了“无缺失值”的模式(所有元素都为1)。第二行表述了“除了Span之外无缺失值”的模式。第一列表示各缺失值模式的实例个数,最后一列表示各模式中有缺失值的变量的个数。此处可以看到,有42个实例没有缺失值,仅2个实例缺失了Span。9个实例同时缺失了NonD和Dream的值。数据集包含了总共(42 × 0) + (2 × 1) + … + (1 × 3) = 38个缺失值。最后一行给出了每个变量中缺失值的数目。
15.3.2 图形探究缺失数据
虽然md.pattern()函数的表格输出非常简洁,但我通常觉得用图形展示模式更为清晰。VIM包提供了大量能可视化数据集中缺失值模式的函数,本节我们将学习其中几个:aggr()、matrixplot()和scattMiss()。
aggr()函数不仅绘制每个变量的缺失值数,还绘制每个变量组合的缺失值数。例如:
library("VIM")aggr(sleep, prop=FALSE, numbers=TRUE)
上述代码的结果见图15-2。(VIM包将会打开GUI界面,你可以关闭它;本章我们使用代码完成所有的工作。)
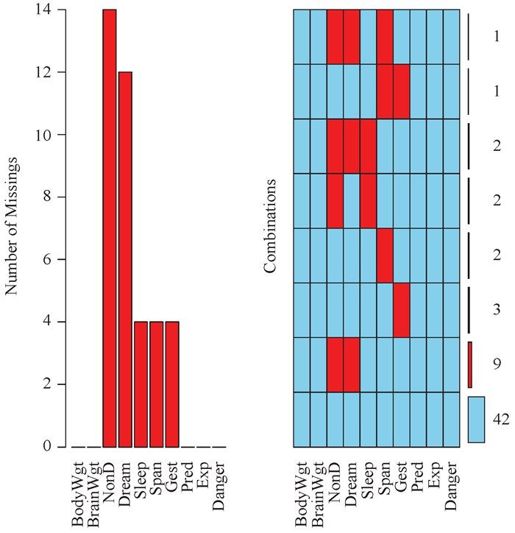
图15-2 aggr()生成的sleep数据集的缺失值模式图形
可以看到,变量NonD有最大的缺失值数(14),有2个哺乳动物缺失了NonD、Dream和Sleep的评分。42个动物没有缺失值。
代码aggr(sleep, prop = TRUE, numbers = TRUE)将生成相同的图形,但用比例代替了计数。选项numbers = FALSE(默认)删去数值型标签。
matrixplot()函数可生成展示每个实例数据的图形。matrixplot(sleep)的图形见图15-3。此处,数值型数据被重新转换到[0, 1]区间,并用灰度来表示大小:浅色表示值小,深色表示值大。默认缺失值为红色。注意,在图15-3中,红色已经被手工阴影化处理,因此相对于灰色缺失值将非常显眼。你可以自己创建图形,让它与众不同。
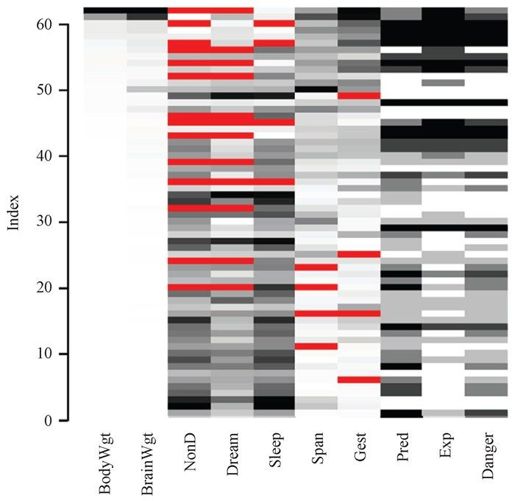
图15-3 sleep数据集按实例(行)展示真实值和缺失值的矩阵图。矩阵按BodyWgt重排
该图形可以进行交互,单击一列将会按其对应的变量重排矩阵。图15-3中的行便按BodyWgt降序排列。通过矩阵图,你可以看出某些变量的缺失值模式是否与其他变量的真实值有关联。此图中可以看到,无缺失值的睡眠变量(Dream、NonD和Sleep)对应着较小的体重(BodyWgt)或脑重(BrainWgt)。
marginplot()函数可生成一幅散点图,在图形边界展示两个变量的缺失值信息。以做梦时长与哺乳动物妊娠期时长的关系为例,来看下列代码:
marginplot(sleep[c("Gest","Dream")], pch=c(20),col=c("darkgray", "red", "blue"))
它的生成图形见图15-4。参数pch和col为可选项,控制着绘图符号和使用的颜色。
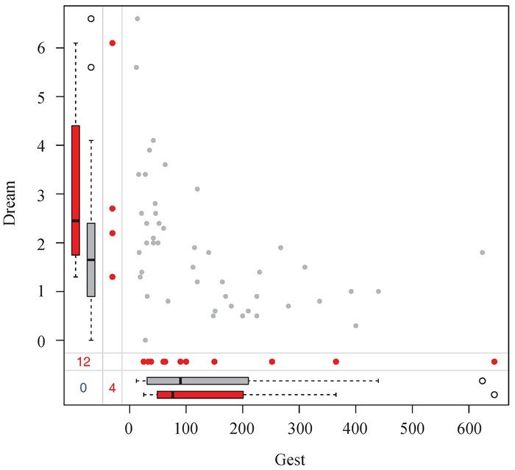
图15-4 做梦时长与妊娠期时长的散点图,边界展示了缺失数据的信息
图形的主体是Gest和Dream(两变量数据都完整)的散点图。左边界的箱线图展示的是包含(深灰色)与不包含(红色)Gest值的Dream变量分布。注意,在灰度图上红色是更深的阴影。四个红色的点代表着缺失了Gest得分的Dream值。在底部边界上,Gest和Dream间的关系反过来了。可以看到,妊娠期和做梦时长呈负相关,缺失妊娠期数据时动物的做梦时长一般更长。两个变量均有缺失值的观测个数在两边界交叉处(左下角)用蓝色输出。
VIM包有许多图形可以帮助你理解缺失数据在数据集中的模式,包括用散点图、箱线图、直方图、散点图矩阵、平行坐标图、轴须图和气泡图来展示缺失值的信息,因此这个包很值得探索。
15.3.3 用相关性探索缺失值
在继续下文之前,还有些方法值得注意。你可用指示变量替代数据集中的数据(1表示缺失,0表示存在),这样生成的矩阵有时称作影子矩阵。求这些指示变量间和它们与初始(可观测)变量间的相关性,有助于观察哪些变量常一起缺失,以及分析变量“缺失”与其他变量间的关系。
考虑如下代码:
x <- as.data.frame(abs(is.na(sleep)))
若sleep的元素缺失,则数据框x对应的元素为1,否则为0。你可以观察下数据的前几行:
> head(sleep, n=5)BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger1 6654.000 5712.0 NA NA 3.3 38.6 645 3 5 32 1.000 6.6 6.3 2.0 8.3 4.5 42 3 1 33 3.385 44.5 NA NA 12.5 14.0 60 1 1 14 0.920 5.7 NA NA 16.5 NA 25 5 2 35 2547.000 4603.0 2.1 1.8 3.9 69.0 624 3 5 4> head(x, n=5)BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger1 0 0 1 1 0 0 0 0 0 02 0 0 0 0 0 0 0 0 0 03 0 0 1 1 0 0 0 0 0 04 0 0 1 1 0 1 0 0 0 05 0 0 0 0 0 0 0 0 0 0
以下代码:
y <- x[which(sd(x) > 0)]
可提取含(但不全部是)缺失值的变量,而
cor(y)
可列出这些指示变量间的相关系数:
NonD Dream Sleep Span GestNonD 1.000 0.907 0.486 0.015 -0.142Dream 0.907 1.000 0.204 0.038 -0.129Sleep 0.486 0.204 1.000 -0.069 -0.069Span 0.015 0.038 -0.069 1.000 0.198Gest -0.142 -0.129 -0.069 0.198 1.000
此时,你可以看到Dream和NonD常常一起缺失(r = 0.91)。相对可能性较小的是Sleep和NonD一起缺失(r = 0.49),以及Sleep和Dream(r = 0.20)。
最后,你可以看到含缺失值变量与其他可观测变量间的关系:
> cor(sleep, y, use="pairwise.complete.obs")NonD Dream Sleep Span GestBodyWgt 0.227 0.223 0.0017 -0.058 -0.054BrainWgt 0.179 0.163 0.0079 -0.079 -0.073NonD NA NA NA -0.043 -0.046Dream -0.189 NA -0.1890 0.117 0.228Sleep -0.080 -0.080 NA 0.096 0.040Span 0.083 0.060 0.0052 NA -0.065Gest 0.202 0.051 0.1597 -0.175 NAPred 0.048 -0.068 0.2025 0.023 -0.201Exp 0.245 0.127 0.2608 -0.193 -0.193Danger 0.065 -0.067 0.2089 -0.067 -0.204Warning message:In cor(sleep, y, use = "pairwise.complete.obs") :the standard deviation is zero
在这个相关系数矩阵中,行为可观测变量,列为表示缺失的指示变量。你可以忽略矩阵中的警告信息和NA值,这些都是方法中人为因素所导致的。
从相关系数矩阵的第一列可以看到,体重越大(r = 0.227)、妊娠期越长(r =0.202)、睡眠暴露度越大(r = 0.245)的动物无梦睡眠的评分更可能缺失。其他列的信息也可以按类似方式得出。注意,表中的相关系数并不特别大,表明数据是MCAR的可能性比较小,更可能为MAR。
不过也绝不能排除数据是NMAR的可能性,因为你并不知道缺失数据背后对应的真实数据是怎么样的。比如,你不可能知道哺乳动物做梦时长与该变量数据缺失概率间的关系。当缺乏强力的外部证据时,我们通常假设数据是MCAR或者MAR。
