D.1 用Sweave(R+LaTeX)实现高质量排版
LaTeX是一个实现高质量排版的文档准备系统(http://www.latex-project.org),在Windows、Mac和Linux平台上都是免费的。作者在文本文件中利用个标记代码实现内容的格式化,然后用LaTeX编译器处理文档,最终得到PDF、PostScript或DVI格式的文档。
利用Sweave包可以将R代码及其输出(包括图形)嵌入到LaTeX文档中。步骤如下。
用文本编辑器创建一个叫做noweb文件的文档(通常后缀是.Rnw)。这个文件中包含报告的内容、LaTeX标记代码和R代码段。每一段R代码都以
<<>>=开头,以@符号结束。Sweave()函数处理noweb文件,生成LaTeX文件。在这一步,R代码会被执行,然后根据设置用符合LaTeX格式的R代码和输出结果替换原来的R代码。这一步可以在R中完成,也可以在命令行中完成。
在R中是这样:
Sweave("infile.Rnw")
默认情况下,Sweave("example.Rnw")会从当前工作目录中读取example.Rnw文件,然后将example.tex文件输出到同一目录中。此外,也可以这样:
Sweave("infile.Rnw", syntax="SweaveSyntaxNoweb")
通过设置syntax选项可以避免一些常见的解析错误,以及跟R2HTML包之间的冲突。
在命令行中进行操作的具体过程取决于所使用的操作系统。例如,在Linux系统上,命令是这样的:
$ R CMD Sweave infile.Rnw
- 然后用LaTeX编译器处理这个LaTeX文件,创建PDF、PostScript或DVI文件。流行的LaTeX编译器有Linux平台上的TeX Live、Mac平台上的MaxTeX和Windows平台上的proTeXt。
图D-1中是整个过程的框架。
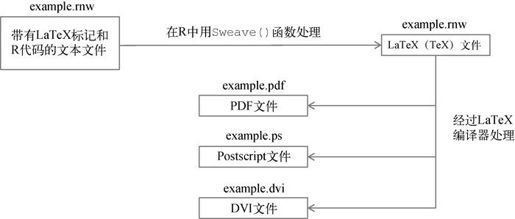
图D-1 用Sweave生成出版质量报告的流程
前面说过,每一段R代码都在<<>>=和@符号中。可以在<<>>=中用各种选项控制相应R代码的处理方法。例如:
<<echo=TRUE, results=HIDE>>=summary(lm(Y~X, data=mydata))@
就只会输出代码,而不会输出结果,而:
<<echo=FALSE, fig=TRUE>>=plot(A)@
就不会显示代码,只会显示输出中的图片。表D-1中列出了常见的选项。 表D-1 R代码块的常见选项
| 选 项 | 描 述 |
|---|---|
echo | 输出中包含代码(echo=TRUE)或不包含(echo=FALSE)。默认是TRUE |
eval | 用eval=FALSE让代码不被求值或运行。默认是TRUE |
fig | 当输出是图形时用fig=TRUE。默认是FALSE |
results | 包含R代码输出(results=verbatim)、不包含输出(results=hide),或者是包含输出并假定其中有LaTeX标记(results=tex)。默认是verbatim。如果输出是用xtable包中的xtable()函数或Hmisc中的latex()函数生成的,就应该用results=tex |
默认情况下,Sweave会添加LaTeX标记代码让数据框、矩阵和向量的格式更加美观。此外,可以用\Sexpr{}语句嵌入R对象。要注意,lattice图形必须嵌入到print()语句中才能正确处理。
xtable包中的xtable()函数可以更精确地格式化数据框和矩阵。此外,它还可以用于格式化其他R对象,包括lm()、glm()、aov()、table()、ts()和coxph()函数生成的对象。用method(xtable)可以看到完整的列表。在用xtable()格式化R输出时,要确保在代码块选项中使用results=tex选项。
看一个例子就明白了。代码清单D-1中是一个noweb文件。这实际上是8.3节中单因子ANOVA分析的例子。LaTeX标记代码是以反斜杠(\)开头的。\Sexpr{}是一个例外,这是一个Sweave的语句。跟R有关的代码用加粗的斜体表示。
代码清单D-1 示例noweb文件(example.nrw)
- \documentclass[12pt]{article}
- \title{Sample Report}
- \author{Robert I. Kabacoff, Ph.D.}
- \date{}
- \begin{document}
- \maketitle
- <<echo=false, results=hide>>=
- library(multcomp)
- library(xtable)
- attach(cholesterol)
- @
- \section{Results}
- Cholesterol reduction was assessed in a study
- that randomized \Sexpr{nrow(cholesterol)} patients
- to one of \Sexpr{length(unique(trt))} treatments.
- Summary statistics are provided in
- Table \ref{table:descriptives}.
- <<echo = false, results = tex>>=
- descTable <- data.frame("Treatment" = sort(unique(trt)),
- "N" = as.vector(table(trt)),
- "Mean" = tapply(response, list(trt), mean, na.rm=TRUE),
- "SD" = tapply(response, list(trt), sd, na.rm=TRUE)
- )
- print(xtable(descTable, caption = "Descriptive statistics
- for each treatment group", label = "table:descriptives"),
- caption.placement = "top", include.rownames = FALSE)
- @
- The analysis of variance is provided in Table \ref{table:anova}.
- <<echo=false, results=tex>>=
- fit <- aov(response ~ trt)
- print(xtable(fit, caption = "Analysis of variance",
- label = "table:anova"), caption.placement = "top")
- @
- \noindent and group differences are plotted in Figure \ref{figure:tukey}.
- \begin{figure}\label{figure:tukey}
- \begin{center}
- <<fig=TRUE,echo=FALSE>>=
- par(mar=c(5,4,6,2))
- tuk <- glht(fit, linfct=mcp(trt="Tukey"))
- plot(cld(tuk, level=.05),col="lightgrey",xlab="Treatment", ylab="Response")
- box("figure")
- @
- \caption{Distribution of response times and pairwise comparisons.}
- \end{center}
- \end{figure}
- \end{document}
在用R中的Sweave()函数处理完noweb文件后,用LaTeX编译器处理所得到的TeX文件,然后会生成图D-2和图D-3所示的PDF文档。
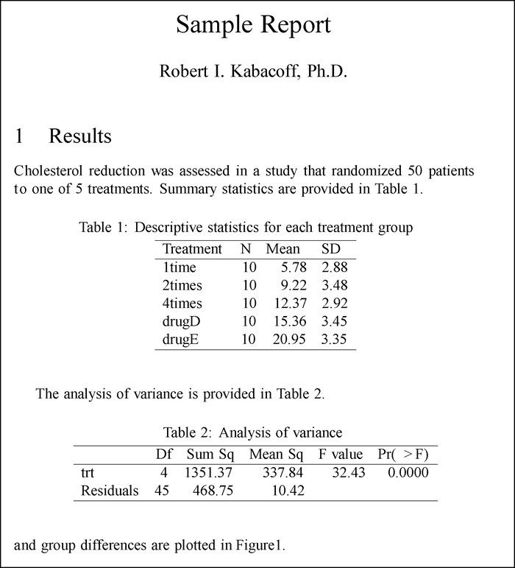
图D-2 从代码清单D-1中的示例noweb文件创建的报告的第一页。在R中用Sweave()函数处理noweb文件,然后用LaTeX编译器处理得到的TeX文件,生成PDF文档
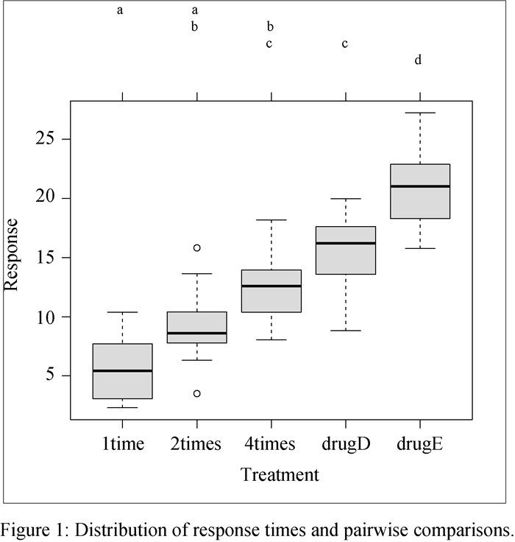
图D-3 用代码清单D-1中的示例noweb文件创建的报告的第二页
关于Sweave的更多信息,请访问Sweave的主页(www.stat.uni-muenchen.de/~leisch/Sweave/)。Theresa Scott提供了一个绝佳的介绍(http://biostat.mc.vanderbilt.edu/TheresaScott)。关于LaTeX的更多信息,可以阅读“The Not So Short Introduction to LaTeX 2e”一文,可在LaTeX主页(www.latex-project.org)上找到。
